Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos

- Dispositivo basado en papel mejora la precisión de prueba del VIH
- Nuevo ensayo LC-MS/MS detecta niveles bajos de creatinina en sudor y saliva
- Avance en biodetección abre camino a nuevos métodos de detección temprana de enfermedades
- Nueva prueba de saliva identifica sobredosis de paracetamol
- Dispositivo de pruebas de saliva predice la insuficiencia cardíaca en 15 minutos
- Simple prueba de orina podría ayudar a evitar exploraciones invasivas para cáncer de riñón
- Nueva prueba para cáncer de intestino mejorará detección temprana
- Prueba refinada mejora diagnóstico de enfermedad de Parkinson
- Nuevo método diagnostica rápidamente riesgo de ECV mediante análisis molecular de sangre
- Análisis de sangre muestra resultados prometedores para detección temprana de demencia
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Sistema de hemostasia POC podría prevenir muertes maternas
- Nueva prueba evalúa capacidad de los glóbulos rojos para transportar oxígeno midiendo su forma
- Prueba de líquido cefalorraquídeo predice efecto secundario peligroso del tratamiento del cáncer
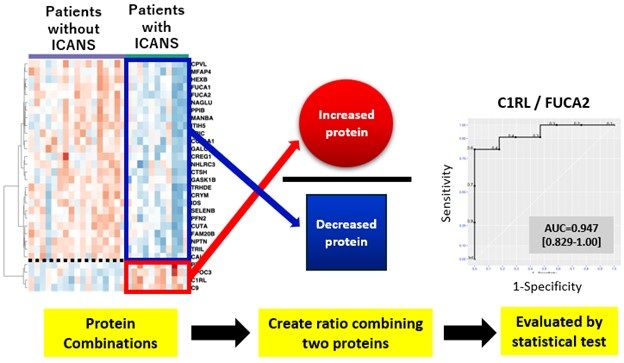
- Nueva prueba mide inmunidad de bebés prematuros usando sólo dos gotas de sangre
- Simple análisis de sangre ayudaría a elegir mejores tratamientos para pacientes con cáncer de endometrio recurrente
- Nuevo método analítico rastrea progresión de enfermedades autoinmunes
- Modelo de cáncer gástrico bioimpreso en 3D utiliza tejido del paciente para predecir respuesta a fármacos
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Avance en impresión 3D permite desarrollo a gran escala de diminutos dispositivos microfluídicos
- Plataforma de sensores en papel transforma diagnóstico cardíaco
- Estudio explora impacto de pruebas POC en el futuro de los diagnósticos
- Sensor económico de respuesta rápida permite detección temprana y precisa del cáncer de pulmón
- Nanotecnología para diagnósticar cáncer de cuello uterino podría sustituir pruebas de Papanicolaou
- Grifols e Inpeco se asocian para crear el "laboratorio del futuro" de medicina transfusional
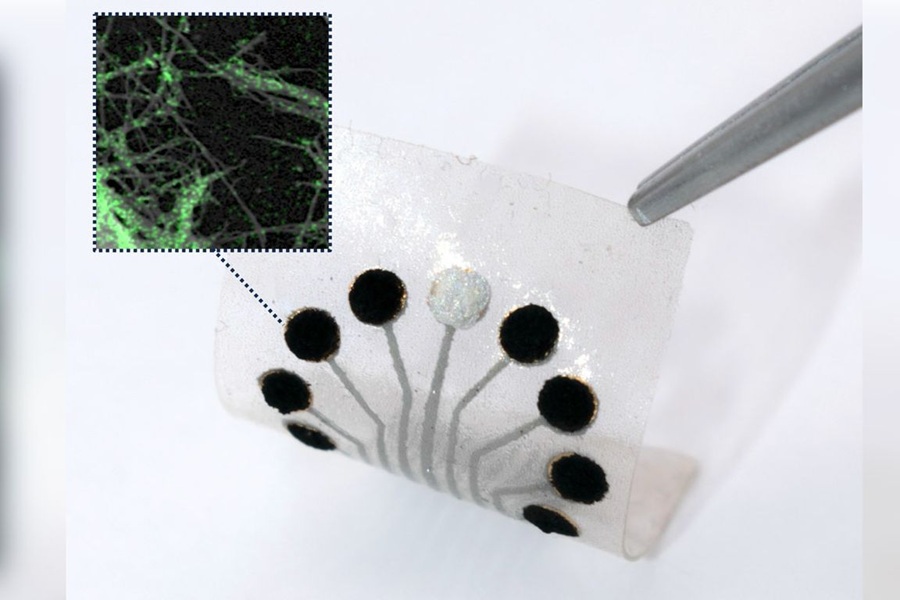
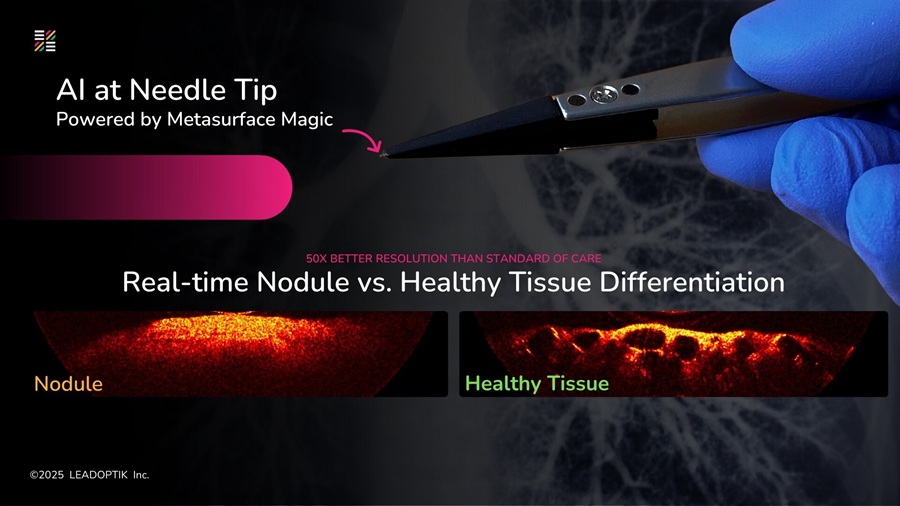
- Investigación impulsa imágenes ópticas en tiempo real mejoradas con IA en biopsias de cáncer de pulmón
- CACLP 2025 reúne innovadores globales de industria del IVD
- Bio-Rad adquirirá Stilla Technologies, desarrollador de PCR digital
- ABL firma acuerdo de licencia y transferencia de know-how para cartera de Fast Track Diagnostics de Siemens
- Condiciones preanalíticas influyen en estabilidad de microARN libres de células en muestras de plasma sanguíneo
- Sistema de cultivo celular 3D podría revolucionar diagnóstico del cáncer
- Técnica indolora mide concentraciones de glucosa en solución y tejido mediante ondas sonoras
- Prueba cutánea mejora diagnóstico de enfermedades neurodegenerativas raras y debilitantes
- Uromodulina sérica podría indicar lesión renal aguda en pacientes con COVID-19

Expo
 ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos
Publique su anuncio con nosotros
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos
Publique su anuncio con nosotros


- Dispositivo basado en papel mejora la precisión de prueba del VIH
- Nuevo ensayo LC-MS/MS detecta niveles bajos de creatinina en sudor y saliva
- Avance en biodetección abre camino a nuevos métodos de detección temprana de enfermedades
- Nueva prueba de saliva identifica sobredosis de paracetamol
- Dispositivo de pruebas de saliva predice la insuficiencia cardíaca en 15 minutos
- Simple prueba de orina podría ayudar a evitar exploraciones invasivas para cáncer de riñón
- Nueva prueba para cáncer de intestino mejorará detección temprana
- Prueba refinada mejora diagnóstico de enfermedad de Parkinson
- Nuevo método diagnostica rápidamente riesgo de ECV mediante análisis molecular de sangre
- Análisis de sangre muestra resultados prometedores para detección temprana de demencia
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Sistema de hemostasia POC podría prevenir muertes maternas
- Nueva prueba evalúa capacidad de los glóbulos rojos para transportar oxígeno midiendo su forma
- Prueba de líquido cefalorraquídeo predice efecto secundario peligroso del tratamiento del cáncer
- Nueva prueba mide inmunidad de bebés prematuros usando sólo dos gotas de sangre
- Simple análisis de sangre ayudaría a elegir mejores tratamientos para pacientes con cáncer de endometrio recurrente
- Nuevo método analítico rastrea progresión de enfermedades autoinmunes
- Modelo de cáncer gástrico bioimpreso en 3D utiliza tejido del paciente para predecir respuesta a fármacos
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Avance en impresión 3D permite desarrollo a gran escala de diminutos dispositivos microfluídicos
- Plataforma de sensores en papel transforma diagnóstico cardíaco
- Estudio explora impacto de pruebas POC en el futuro de los diagnósticos
- Sensor económico de respuesta rápida permite detección temprana y precisa del cáncer de pulmón
- Nanotecnología para diagnósticar cáncer de cuello uterino podría sustituir pruebas de Papanicolaou
- Grifols e Inpeco se asocian para crear el "laboratorio del futuro" de medicina transfusional
- Investigación impulsa imágenes ópticas en tiempo real mejoradas con IA en biopsias de cáncer de pulmón
- CACLP 2025 reúne innovadores globales de industria del IVD
- Bio-Rad adquirirá Stilla Technologies, desarrollador de PCR digital
- ABL firma acuerdo de licencia y transferencia de know-how para cartera de Fast Track Diagnostics de Siemens
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Condiciones preanalíticas influyen en estabilidad de microARN libres de células en muestras de plasma sanguíneo
- Sistema de cultivo celular 3D podría revolucionar diagnóstico del cáncer
- Técnica indolora mide concentraciones de glucosa en solución y tejido mediante ondas sonoras
- Prueba cutánea mejora diagnóstico de enfermedades neurodegenerativas raras y debilitantes
- Uromodulina sérica podría indicar lesión renal aguda en pacientes con COVID-19