Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos

- Dispositivo basado en papel mejora la precisión de prueba del VIH
- Nuevo ensayo LC-MS/MS detecta niveles bajos de creatinina en sudor y saliva
- Avance en biodetección abre camino a nuevos métodos de detección temprana de enfermedades
- Nueva prueba de saliva identifica sobredosis de paracetamol
- Dispositivo de pruebas de saliva predice la insuficiencia cardíaca en 15 minutos
- Nuevos biomarcadores mejoran la detección temprana y seguimiento de la lesión renal
- Inmunoensayos de quimioluminiscencia respaldan diagnóstico de Alzheimer
- Análisis de sangre identifica múltiples biomarcadores para diagnóstico rápido de lesiones de médula espinal
- Análisis de sangre muy preciso diagnostica Alzheimer y mide progresión de demencia
- Prueba sencilla basada en PCR de ADN permite tratamiento personalizado de vaginosis bacteriana
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Sistema de hemostasia POC podría prevenir muertes maternas
- Nueva prueba evalúa capacidad de los glóbulos rojos para transportar oxígeno midiendo su forma
- Prueba de líquido cefalorraquídeo predice efecto secundario peligroso del tratamiento del cáncer
- Nueva prueba mide inmunidad de bebés prematuros usando sólo dos gotas de sangre
- Simple análisis de sangre ayudaría a elegir mejores tratamientos para pacientes con cáncer de endometrio recurrente
- Nuevo método analítico rastrea progresión de enfermedades autoinmunes
- Modelo de cáncer gástrico bioimpreso en 3D utiliza tejido del paciente para predecir respuesta a fármacos
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
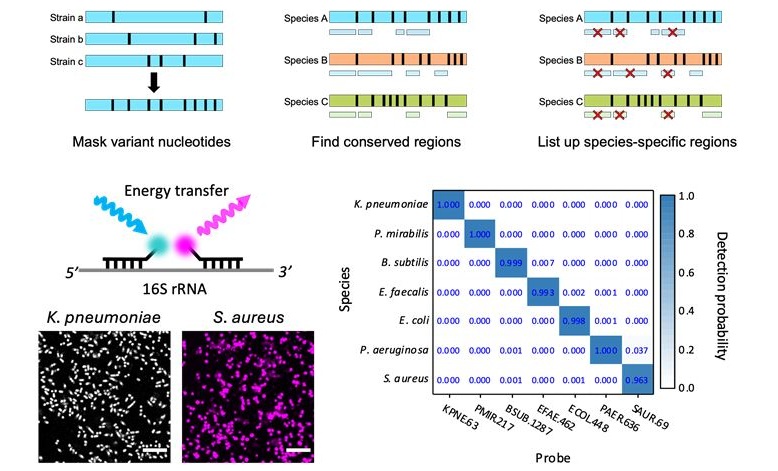
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Nueva tecnología de sensores permite diagnóstico temprano de trastornos metabólicos y cardiovasculares
- Avance en impresión 3D permite desarrollo a gran escala de diminutos dispositivos microfluídicos
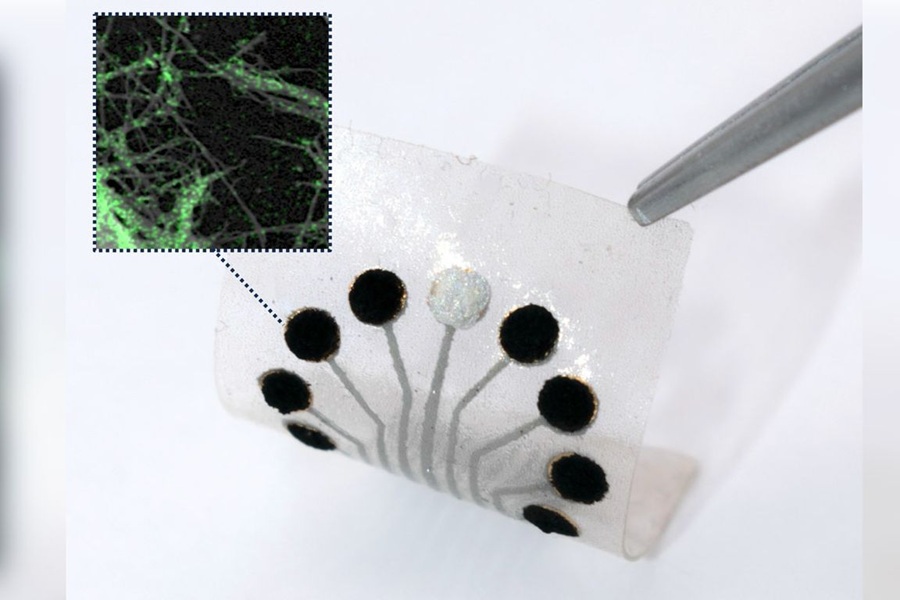
- Plataforma de sensores en papel transforma diagnóstico cardíaco
- Estudio explora impacto de pruebas POC en el futuro de los diagnósticos
- Sensor económico de respuesta rápida permite detección temprana y precisa del cáncer de pulmón
- Leica Biosystems y Bio-Techne amplían su colaboración multiómica espacial
- Philips e Ibex amplían colaboración para mejorar flujos de trabajo de patología basados en IA
- Grifols e Inpeco se asocian para crear el "laboratorio del futuro" de medicina transfusional
- Investigación impulsa imágenes ópticas en tiempo real mejoradas con IA en biopsias de cáncer de pulmón
- CACLP 2025 reúne innovadores globales de industria del IVD
- Nuevo método basado en láser acelera diagnóstico del cáncer
- Nuevo modelo de IA predice efectos de variantes genéticas en enfermedades específicas
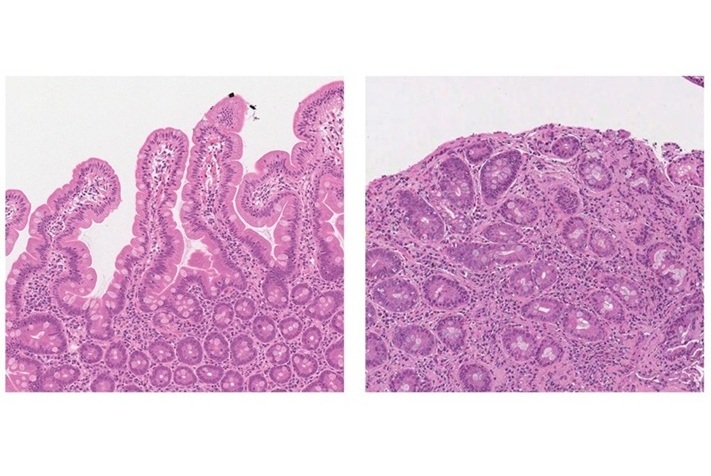
- Herramienta de IA diagnostica enfermedad celíaca en imágenes de biopsia con precisión superior al 97%
- Condiciones preanalíticas influyen en estabilidad de microARN libres de células en muestras de plasma sanguíneo
- Sistema de cultivo celular 3D podría revolucionar diagnóstico del cáncer

Expo
 ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos
Publique su anuncio con nosotros
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos
Publique su anuncio con nosotros


- Dispositivo basado en papel mejora la precisión de prueba del VIH
- Nuevo ensayo LC-MS/MS detecta niveles bajos de creatinina en sudor y saliva
- Avance en biodetección abre camino a nuevos métodos de detección temprana de enfermedades
- Nueva prueba de saliva identifica sobredosis de paracetamol
- Dispositivo de pruebas de saliva predice la insuficiencia cardíaca en 15 minutos
- Nuevos biomarcadores mejoran la detección temprana y seguimiento de la lesión renal
- Inmunoensayos de quimioluminiscencia respaldan diagnóstico de Alzheimer
- Análisis de sangre identifica múltiples biomarcadores para diagnóstico rápido de lesiones de médula espinal
- Análisis de sangre muy preciso diagnostica Alzheimer y mide progresión de demencia
- Prueba sencilla basada en PCR de ADN permite tratamiento personalizado de vaginosis bacteriana
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Sistema de hemostasia POC podría prevenir muertes maternas
- Nueva prueba evalúa capacidad de los glóbulos rojos para transportar oxígeno midiendo su forma
- Prueba de líquido cefalorraquídeo predice efecto secundario peligroso del tratamiento del cáncer
- Nueva prueba mide inmunidad de bebés prematuros usando sólo dos gotas de sangre
- Simple análisis de sangre ayudaría a elegir mejores tratamientos para pacientes con cáncer de endometrio recurrente
- Nuevo método analítico rastrea progresión de enfermedades autoinmunes
- Modelo de cáncer gástrico bioimpreso en 3D utiliza tejido del paciente para predecir respuesta a fármacos
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Nueva tecnología de sensores permite diagnóstico temprano de trastornos metabólicos y cardiovasculares
- Avance en impresión 3D permite desarrollo a gran escala de diminutos dispositivos microfluídicos
- Plataforma de sensores en papel transforma diagnóstico cardíaco
- Estudio explora impacto de pruebas POC en el futuro de los diagnósticos
- Sensor económico de respuesta rápida permite detección temprana y precisa del cáncer de pulmón
- Leica Biosystems y Bio-Techne amplían su colaboración multiómica espacial
- Philips e Ibex amplían colaboración para mejorar flujos de trabajo de patología basados en IA
- Grifols e Inpeco se asocian para crear el "laboratorio del futuro" de medicina transfusional
- Investigación impulsa imágenes ópticas en tiempo real mejoradas con IA en biopsias de cáncer de pulmón
- CACLP 2025 reúne innovadores globales de industria del IVD
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Nuevo método basado en láser acelera diagnóstico del cáncer
- Nuevo modelo de IA predice efectos de variantes genéticas en enfermedades específicas
- Herramienta de IA diagnostica enfermedad celíaca en imágenes de biopsia con precisión superior al 97%
- Condiciones preanalíticas influyen en estabilidad de microARN libres de células en muestras de plasma sanguíneo
- Sistema de cultivo celular 3D podría revolucionar diagnóstico del cáncer