Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunologíaMicrobiologíaPatología
Industria
Eventos

- Herramienta de diagnóstico identifica múltiples condiciones de salud a partir de una sola gota de sangre
- Un analizador integrado de química e inmunoensayo con extenso menú de ensayos ofrece flexibilidad, escalabilidad y conmutabilidad de datos
- Prueba rápida de fármacos podría mejorar el tratamiento de pacientes que se presentan en el hospital
- Modelo de IA detecta el cáncer a una velocidad relámpago mediante análisis de azúcar
- El primer chip alimentado por sangre ofrece monitoreo de salud en tiempo real
- Análisis de sangre más inteligentes permiten diagnósticos más rápidos y mejores resultados
- Una única prueba de secuenciación metagenómica de próxima generación detecta patógenos infecciosos
- Herramienta de diagnóstico identifica rápidamente variantes emergentes del SARS-CoV-2
- Nuevo método analiza variaciones genéticas en familias con alta incidencia de cáncer de mama
- Prueba de ADNlc reduce riesgos del embarazo
- Sistema de diagnóstico de hemograma completo y sepsis busca resultados más rápidos, tempranos y fáciles
- Nuevo grupo sanguíneo descubierto ayudará a identificar y tratar a pacientes
- Puntuación de plaquetas sanguíneas detecta el riesgo de ataque cardíaco y accidente cerebrovascular
- Sistema automatizado de sobremesa lleva pruebas de sangre a cualquier persona, en cualquier lugar
- Nuevos analizadores de hematología ofrecen resultados combinados de VSG y CBC/DIFF en 60 segundos
- Método de imágenes mapea conexiones entre células inmunes para predecir supervivencia de pacientes con cáncer
- Herramienta computacional predice resultados de inmunoterapia en cáncer de mama metastásico
- Biomarcador podría predecir respuesta a inmunoterapia en cáncer de hígado
- Prueba epigenética podría determinar eficacia de nuevos tratamientos de inmunoterapia contra mieloma múltiple
- Análisis de sangre predice la supervivencia en pacientes con cáncer de hígado
- Prueba de cabecera de alta precisión diagnostica infección articular periprotésica en cinco minutos
- Enfoque innovador para diagnóstico de infecciones bacterianas permitirá un tratamiento más rápido y eficaz
- Análisis de heces diagnostica endometriosis y ayuda a reducir progresión de la enfermedad
- Plataforma de preparación de muestras de hemocultivos positivos diseñada para combatir sepsis y resistencia a antimicrobianos
- Tecnología revolucionaria de detección molecular podría transformar el diagnóstico de enfermedades infecciosas
- Tecnología de diagnóstico basada en nanomateriales controla farmacoterapia en pacientes epilépticos
- Métodos no invasivos detectan exposición al plomo de forma más rápida, sencilla y precisa en POC
- Prueba no invasiva detecta malaria sin muestra sanguínea
- Dispositivo portátil detecta cáncer colorrectal y de próstata en una hora
- Tecnología Light-AI para diagnóstico del cáncer eliminaría necesidad de extracciones de sangre y biopsias tradicionales
- Microbiologics adquiere fabricante de controles de calidad de diagnóstico SensID
- Beckman Coulter se asocia con BioPorto para distribución global de pruebas NGAL para lesiones renales agudas
- Se anuncia nueva fecha y sede del CACLP 2025
- Roche desarrollará nuevas tecnologías de diagnóstico para lesiones cerebrales traumáticas
- LGC Clinical Diagnostics y AccuGenomics colaboran para mejorar precisión de pruebas de cáncer
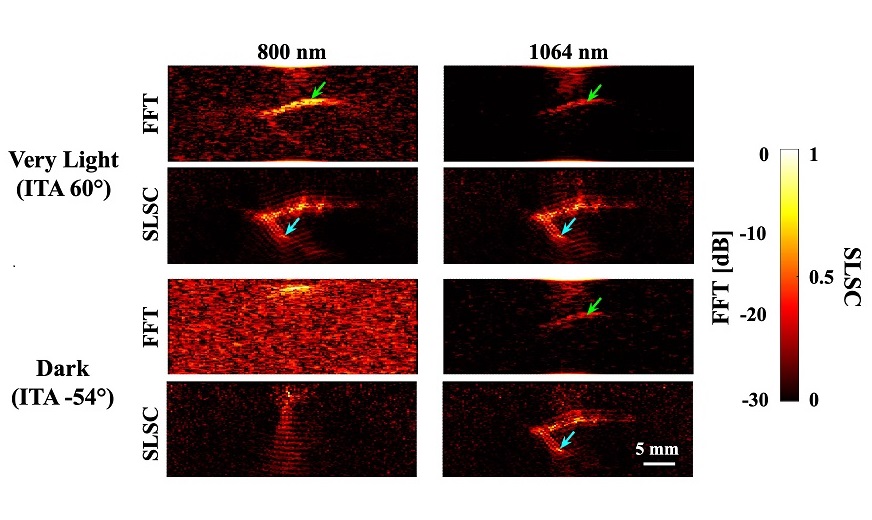
- Técnica de imagenología reduce sesgo del tono de piel en detección del cáncer mamario
- Herramienta de IA "ve" firmas genéticas del cáncer en imágenes de biopsias
- Herramienta de IA identifica nuevas firmas genéticas para personalizar terapias contra cáncer
- Herramienta de IA detecta tumor cerebral canceroso durante cirugía en 10 segundos
- Analizador de imágenes de portaobjetos completo con IA predice respuesta a inmunoterapia en cáncer poco frecuente

Expo
 ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunologíaMicrobiologíaPatología
Industria
Eventos
Publique su anuncio con nosotros
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunologíaMicrobiologíaPatología
Industria
Eventos
Publique su anuncio con nosotros


- Herramienta de diagnóstico identifica múltiples condiciones de salud a partir de una sola gota de sangre
- Un analizador integrado de química e inmunoensayo con extenso menú de ensayos ofrece flexibilidad, escalabilidad y conmutabilidad de datos
- Prueba rápida de fármacos podría mejorar el tratamiento de pacientes que se presentan en el hospital
- Modelo de IA detecta el cáncer a una velocidad relámpago mediante análisis de azúcar
- El primer chip alimentado por sangre ofrece monitoreo de salud en tiempo real
- Análisis de sangre más inteligentes permiten diagnósticos más rápidos y mejores resultados
- Una única prueba de secuenciación metagenómica de próxima generación detecta patógenos infecciosos
- Herramienta de diagnóstico identifica rápidamente variantes emergentes del SARS-CoV-2
- Nuevo método analiza variaciones genéticas en familias con alta incidencia de cáncer de mama
- Prueba de ADNlc reduce riesgos del embarazo
- Sistema de diagnóstico de hemograma completo y sepsis busca resultados más rápidos, tempranos y fáciles
- Nuevo grupo sanguíneo descubierto ayudará a identificar y tratar a pacientes
- Puntuación de plaquetas sanguíneas detecta el riesgo de ataque cardíaco y accidente cerebrovascular
- Sistema automatizado de sobremesa lleva pruebas de sangre a cualquier persona, en cualquier lugar
- Nuevos analizadores de hematología ofrecen resultados combinados de VSG y CBC/DIFF en 60 segundos
- Método de imágenes mapea conexiones entre células inmunes para predecir supervivencia de pacientes con cáncer
- Herramienta computacional predice resultados de inmunoterapia en cáncer de mama metastásico
- Biomarcador podría predecir respuesta a inmunoterapia en cáncer de hígado
- Prueba epigenética podría determinar eficacia de nuevos tratamientos de inmunoterapia contra mieloma múltiple
- Análisis de sangre predice la supervivencia en pacientes con cáncer de hígado
- Prueba de cabecera de alta precisión diagnostica infección articular periprotésica en cinco minutos
- Enfoque innovador para diagnóstico de infecciones bacterianas permitirá un tratamiento más rápido y eficaz
- Análisis de heces diagnostica endometriosis y ayuda a reducir progresión de la enfermedad
- Plataforma de preparación de muestras de hemocultivos positivos diseñada para combatir sepsis y resistencia a antimicrobianos
- Tecnología revolucionaria de detección molecular podría transformar el diagnóstico de enfermedades infecciosas
- Tecnología de diagnóstico basada en nanomateriales controla farmacoterapia en pacientes epilépticos
- Métodos no invasivos detectan exposición al plomo de forma más rápida, sencilla y precisa en POC
- Prueba no invasiva detecta malaria sin muestra sanguínea
- Dispositivo portátil detecta cáncer colorrectal y de próstata en una hora
- Tecnología Light-AI para diagnóstico del cáncer eliminaría necesidad de extracciones de sangre y biopsias tradicionales
- Microbiologics adquiere fabricante de controles de calidad de diagnóstico SensID
- Beckman Coulter se asocia con BioPorto para distribución global de pruebas NGAL para lesiones renales agudas
- Se anuncia nueva fecha y sede del CACLP 2025
- Roche desarrollará nuevas tecnologías de diagnóstico para lesiones cerebrales traumáticas
- LGC Clinical Diagnostics y AccuGenomics colaboran para mejorar precisión de pruebas de cáncer
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Técnica de imagenología reduce sesgo del tono de piel en detección del cáncer mamario
- Herramienta de IA "ve" firmas genéticas del cáncer en imágenes de biopsias
- Herramienta de IA identifica nuevas firmas genéticas para personalizar terapias contra cáncer
- Herramienta de IA detecta tumor cerebral canceroso durante cirugía en 10 segundos
- Analizador de imágenes de portaobjetos completo con IA predice respuesta a inmunoterapia en cáncer poco frecuente


















.jpg)
.jpg)
 (1).jpg)
_1_1.jpg)
_1.jpg)
.jpg)




_1.jpg)




