Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química Clínica
HematologíaInmunologíaMicrobiologíaPatologíaTecnologíaIndustria
Eventos

- Prueba de detección portátil económica transforma detección de enfermedades renales
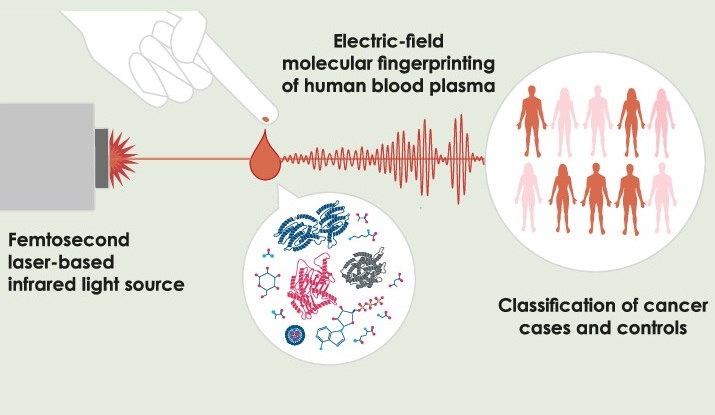
- Nuevo método utiliza luz infrarroja pulsada para encontrar huellas del cáncer en plasma sanguíneo
- Nanotubos de carbono ayudan a construir sensores precisos para monitoreo continuo de la salud
- Dispositivo basado en papel mejora la precisión de prueba del VIH
- Nuevo ensayo LC-MS/MS detecta niveles bajos de creatinina en sudor y saliva
- Se descubre nuevo autoanticuerpo contra DAGLA en cerebelitis
- Análisis de sangre podría identificar a pacientes con riesgo de esclerodermia grave
- Prueba de sangre basada en genes predice recurrencia del cáncer de piel avanzado
- Prueba de sangre rápida identifica pacientes presintomáticos con enfermedad de Parkinson
- Análisis de sangre para detección temprana del Alzheimer con precisión de 90 %
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Sistema de hemostasia POC podría prevenir muertes maternas
- Nueva prueba evalúa capacidad de los glóbulos rojos para transportar oxígeno midiendo su forma
- Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
- Análisis de sangre podría orientar decisiones futuras sobre tratamiento del cáncer
- Prueba de líquido cefalorraquídeo predice efecto secundario peligroso del tratamiento del cáncer
- Nueva prueba mide inmunidad de bebés prematuros usando sólo dos gotas de sangre
- Simple análisis de sangre ayudaría a elegir mejores tratamientos para pacientes con cáncer de endometrio recurrente
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
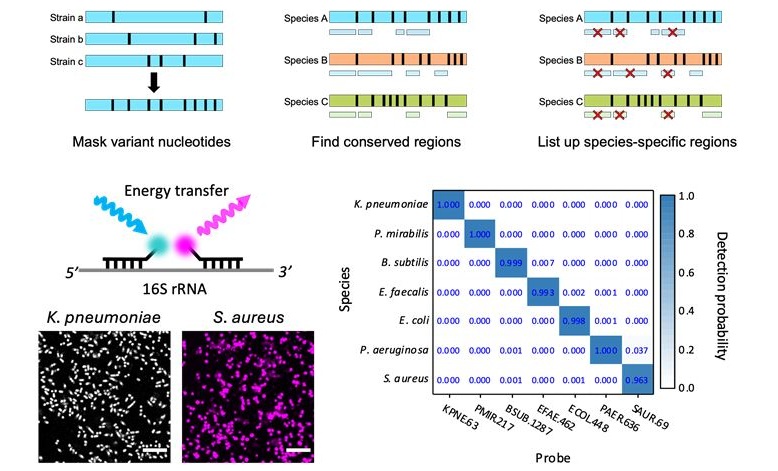
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Dispositivo microfluídico Dolor en un Chip determina tipos de dolor crónico desde muestras de sangre
- Innovador sensor fluorométrico sin etiquetas permite detección más sensible del ARN viral
- Teléfonos inteligentes podrían diagnosticar enfermedades mediante escáneres infrarrojos
- Nueva tecnología de sensores permite diagnóstico temprano de trastornos metabólicos y cardiovasculares
- Avance en impresión 3D permite desarrollo a gran escala de diminutos dispositivos microfluídicos
- Grifols e IBL de Tecan colaboran en paneles de biomarcadores avanzados
- Nueva colaboración avanza en identificación microbiana para diagnóstico de enfermedades infecciosas
- Tecan adquiere activos de inmunoensayo ELISA de Cisbio Bioassays de Revvity
- Leica Biosystems y Bio-Techne amplían su colaboración multiómica espacial
- Philips e Ibex amplían colaboración para mejorar flujos de trabajo de patología basados en IA
- Nuevo método con corrección de errores detecta cáncer únicamente en muestras de sangre
- Algoritmo "detector de metales" consigue tumores vulnerables
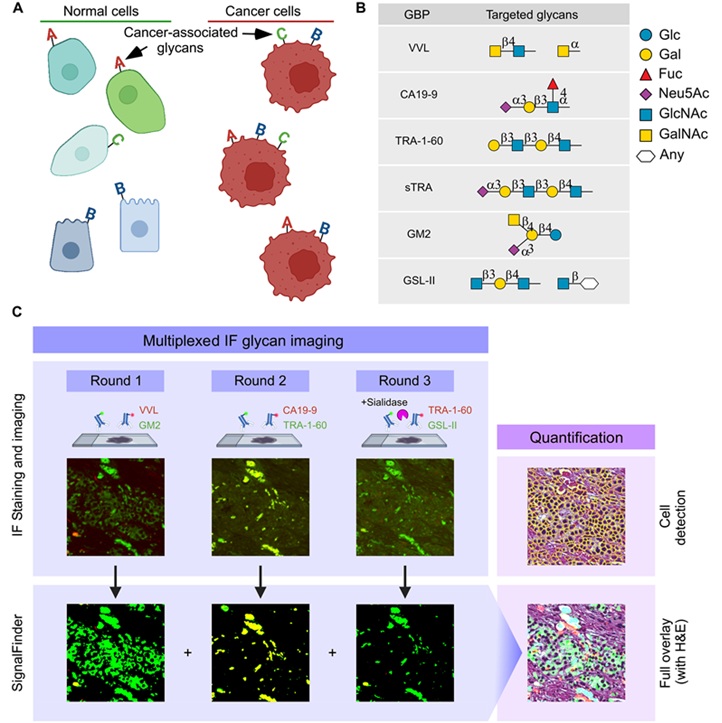
- Nueva técnica identifica y clasifica subtipos de células de cáncer de páncreas
- Imágenes avanzadas revelan mecanismos que causan enfermedades autoinmunes
- Modelo de IA predice eficazmente resultados de pacientes con cáncer de pulmón

 Expo
Expo
- Prueba de detección portátil económica transforma detección de enfermedades renales
- Nuevo método utiliza luz infrarroja pulsada para encontrar huellas del cáncer en plasma sanguíneo
- Nanotubos de carbono ayudan a construir sensores precisos para monitoreo continuo de la salud
- Dispositivo basado en papel mejora la precisión de prueba del VIH
- Nuevo ensayo LC-MS/MS detecta niveles bajos de creatinina en sudor y saliva
- Se descubre nuevo autoanticuerpo contra DAGLA en cerebelitis
- Análisis de sangre podría identificar a pacientes con riesgo de esclerodermia grave
- Prueba de sangre basada en genes predice recurrencia del cáncer de piel avanzado
- Prueba de sangre rápida identifica pacientes presintomáticos con enfermedad de Parkinson
- Análisis de sangre para detección temprana del Alzheimer con precisión de 90 %
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Sistema de hemostasia POC podría prevenir muertes maternas
- Nueva prueba evalúa capacidad de los glóbulos rojos para transportar oxígeno midiendo su forma
- Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
- Análisis de sangre podría orientar decisiones futuras sobre tratamiento del cáncer
- Prueba de líquido cefalorraquídeo predice efecto secundario peligroso del tratamiento del cáncer
- Nueva prueba mide inmunidad de bebés prematuros usando sólo dos gotas de sangre
- Simple análisis de sangre ayudaría a elegir mejores tratamientos para pacientes con cáncer de endometrio recurrente
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Dispositivo microfluídico Dolor en un Chip determina tipos de dolor crónico desde muestras de sangre
- Innovador sensor fluorométrico sin etiquetas permite detección más sensible del ARN viral
- Teléfonos inteligentes podrían diagnosticar enfermedades mediante escáneres infrarrojos
- Nueva tecnología de sensores permite diagnóstico temprano de trastornos metabólicos y cardiovasculares
- Avance en impresión 3D permite desarrollo a gran escala de diminutos dispositivos microfluídicos
- Grifols e IBL de Tecan colaboran en paneles de biomarcadores avanzados
- Nueva colaboración avanza en identificación microbiana para diagnóstico de enfermedades infecciosas
- Tecan adquiere activos de inmunoensayo ELISA de Cisbio Bioassays de Revvity
- Leica Biosystems y Bio-Techne amplían su colaboración multiómica espacial
- Philips e Ibex amplían colaboración para mejorar flujos de trabajo de patología basados en IA
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Nuevo método con corrección de errores detecta cáncer únicamente en muestras de sangre
- Algoritmo "detector de metales" consigue tumores vulnerables
- Nueva técnica identifica y clasifica subtipos de células de cáncer de páncreas
- Imágenes avanzadas revelan mecanismos que causan enfermedades autoinmunes
- Modelo de IA predice eficazmente resultados de pacientes con cáncer de pulmón








.jpg)










.jpg)