Expo
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
ver canal
Química ClínicaDiagnóstico MolecularHematologíaInmunología
PatologíaTecnologíaIndustria
Eventos
Webinars

- Herramienta tipo bolígrafo rápida y no invasiva detecta opioides en piel
- Simple prueba de orina podría detectar múltiples cánceres en etapa temprana
- Prueba de cerumen detecta con precisión Parkinson identificando moléculas de olor
- Método cuantitativo evalúa exposición a opioides en recién nacidos
- Dispositivos basados en papel mejoran métodos existentes en diagnóstico de malaria asintomática
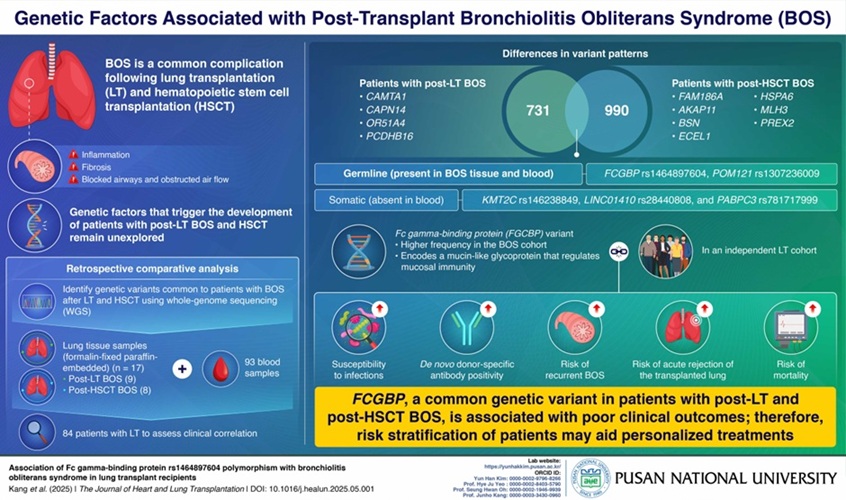
- Prueba genética podría predecir malos resultados en pacientes con trasplante de pulmón
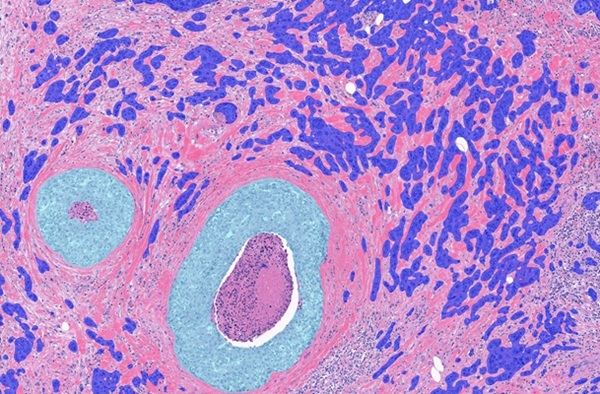
- Análisis de sangre permite detección temprana del cáncer de páncreas
- Pruebas genómicas en la UCIN reducen diagnósticos erróneos
- Nueva prueba genética mejora predicción y clasificación de diabetes
- Prueba de sangre para riesgo de leucemia reemplazaría toma de muestras de médula ósea
- Primera prueba de monitorización de heparina POC proporciona resultados rápidos
- Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Enfoque de biopsia líquida transforma diagnóstico, seguimiento y tratamiento del cáncer de pulmón
- Herramienta computacional expone cambios en ADN canceroso que influyen en resistencia al tratamiento
- Nueva herramienta detecta recaídas de cáncer de mama cinco años antes
- Células T en la sangre pueden detectar Parkinson años antes del diagnóstico
- Plataforma de diagnóstico POC realiza análisis inmunológico con una gota de sangre del dedo
- Perfil de metabolitos fecales predice mortalidad en pacientes críticos
- Sistema portátil de análisis molecular POC descarta infecciones urinarias en 35 minutos
- Prueba de flujo lateral POC detecta infección fúngica mortal más rápido que técnicas existentes
- Prueba de diagnóstico rápido reduce mortalidad por sepsis 39 %
- Análisis de hemocultivo mejora gestión diagnóstica mediante selección de panel específico
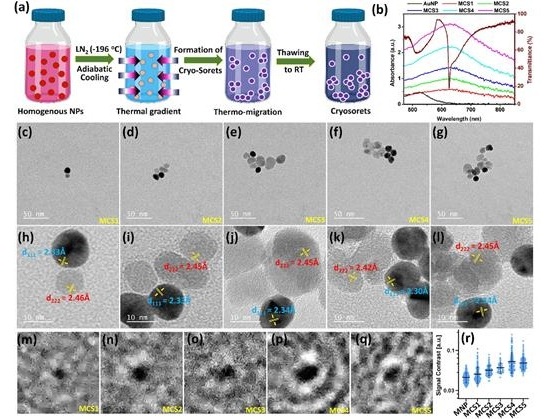
- Avances en biodetección para detección temprana de biomarcadores de enfermedades POC
- Nueva tecnología de biodetección POC mejora detección de biomarcadores moleculares
- Encuesta revela que gestión mejorada de datos de laboratorio e IA son cruciales para laboratorios del futuro
- Prueba no basada en ADN con IA identifica infecciones virales
- Método de IA predice tasa de supervivencia de pacientes con cáncer de próstata
- Illumina adquiere SomaLogic y acelera su negocio de proteómica
- Qiagen y Gencurix se asocian para desarrollar el ensayo de PCR IVD digital QIAcuity
- QIAGEN e Incyte firman acuerdo de colaboración en medicina de precisión
- bioMérieux adquiere soluciones y tecnologías de diagnóstico Day Zero
- Universidad de Aston y BG Research se asocian para comercializar diagnóstico médico innovador
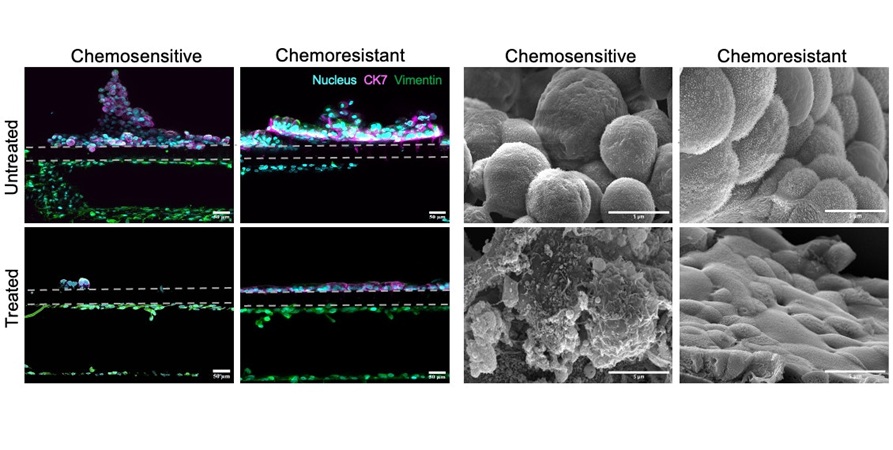
- Chip oncológico predice respuesta a quimioterapia específica del paciente
- Solución de IA clínica para clasificación automática del cáncer de mama mejora precisión diagnóstica
- Pruebas basadas en saliva permiten detección temprana del cáncer, enfermedades cardíacas o Parkinson
- Avances en diagnóstico del virus de viruela del simio mejora gestión de brotes
- Parche con nanoagujas podría eliminar biopsias dolorosas e invasivas

 Expo
Expo
- Herramienta tipo bolígrafo rápida y no invasiva detecta opioides en piel
- Simple prueba de orina podría detectar múltiples cánceres en etapa temprana
- Prueba de cerumen detecta con precisión Parkinson identificando moléculas de olor
- Método cuantitativo evalúa exposición a opioides en recién nacidos
- Dispositivos basados en papel mejoran métodos existentes en diagnóstico de malaria asintomática
- Prueba genética podría predecir malos resultados en pacientes con trasplante de pulmón
- Análisis de sangre permite detección temprana del cáncer de páncreas
- Pruebas genómicas en la UCIN reducen diagnósticos erróneos
- Nueva prueba genética mejora predicción y clasificación de diabetes
- Prueba de sangre para riesgo de leucemia reemplazaría toma de muestras de médula ósea
- Primera prueba de monitorización de heparina POC proporciona resultados rápidos
- Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
- Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
- Recuento de leucocitos predice gravedad de síntomas de COVID-19
- Tecnología de recuento de plaquetas ayudará a prevenir errores de diagnóstico
- Enfoque de biopsia líquida transforma diagnóstico, seguimiento y tratamiento del cáncer de pulmón
- Herramienta computacional expone cambios en ADN canceroso que influyen en resistencia al tratamiento
- Nueva herramienta detecta recaídas de cáncer de mama cinco años antes
- Células T en la sangre pueden detectar Parkinson años antes del diagnóstico
- Plataforma de diagnóstico POC realiza análisis inmunológico con una gota de sangre del dedo
- Perfil de metabolitos fecales predice mortalidad en pacientes críticos
- Sistema portátil de análisis molecular POC descarta infecciones urinarias en 35 minutos
- Prueba de flujo lateral POC detecta infección fúngica mortal más rápido que técnicas existentes
- Prueba de diagnóstico rápido reduce mortalidad por sepsis 39 %
- Análisis de hemocultivo mejora gestión diagnóstica mediante selección de panel específico
- Avances en biodetección para detección temprana de biomarcadores de enfermedades POC
- Nueva tecnología de biodetección POC mejora detección de biomarcadores moleculares
- Encuesta revela que gestión mejorada de datos de laboratorio e IA son cruciales para laboratorios del futuro
- Prueba no basada en ADN con IA identifica infecciones virales
- Método de IA predice tasa de supervivencia de pacientes con cáncer de próstata
- Illumina adquiere SomaLogic y acelera su negocio de proteómica
- Qiagen y Gencurix se asocian para desarrollar el ensayo de PCR IVD digital QIAcuity
- QIAGEN e Incyte firman acuerdo de colaboración en medicina de precisión
- bioMérieux adquiere soluciones y tecnologías de diagnóstico Day Zero
- Universidad de Aston y BG Research se asocian para comercializar diagnóstico médico innovador
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Chip oncológico predice respuesta a quimioterapia específica del paciente
- Solución de IA clínica para clasificación automática del cáncer de mama mejora precisión diagnóstica
- Pruebas basadas en saliva permiten detección temprana del cáncer, enfermedades cardíacas o Parkinson
- Avances en diagnóstico del virus de viruela del simio mejora gestión de brotes
- Parche con nanoagujas podría eliminar biopsias dolorosas e invasivas








.jpg)